Глава 8. Хранение Больших данных
Содержание
Данная глава рассматривает
-
Знакомство с fsspec, библиотекой уровня абстракций поверх файловых систем
-
Эффективное хранение разнородных представляемых столбцами данных при помощи Parquet
-
Обработку файлов данных при помощи библиотек работы в оперативной памяти, таких как pandas или Parquet
-
Обработка однородного многомерного массива при помощи Zarr
При работе с Большими данными постоянное хранение имеет первостепенное значение. Мы хотим обладать возможностью доступа - считывания и записи - к данным как можно более быстрым образом, причём предпочтительно из множества одновременных процессов. Нам также требуются компактные представления постоянного хранения, поскольку хранение больших объёмов данных может быть дорогостоящим.
В данной главе мы рассмотрим несколько подходов к повышению эффективности постоянного хранения данных. Начнём мы с краткого обсуждения fsspec, библиотеки, которая представляет уровень абстракции доступа к файловым системам, причём как к локальным, так и к удалённым. Хотя fsspec напрямую не связана с задачами производительности, это современная библиотека для работы с системами хранения и она часто применяется в действенных реализациях хранения.
Затем мы рассмотрим Parquet, файловый формат для постоянного хранения неоднородных наборов данных в форме столбцов. Parquet поддерживается в Python посредством проекта Apache Arrow, который был представлен в нашей предыдущей главе.
После этого мы обсудим считывание фрагментами очень больших наборов данных, порой именуемого подходом out-of-core (не умещающихся в оперативной памяти данных, вне быстрой памяти). Зачастую у нас имеются хранимые наборы данных, которые невозможно обрабатывать в оперативной памяти одновременно. Считывание фрагментами позволяет вам обрабатывать по частям с применением уже знакомых вам библиотек, что является простой, но очень действенной стратегией. Наш пример возьмёт большой кадр данных pandas и преобразует его в файл Parquet. Наконец, мы рассмотрим Zarr, современный формат и библиотеку постоянного хранения многомерных однородных массивов (то есть массивов NumPy).
Для этой главы вам потребуется установить fsspec, Zarr и Arrow, которые предоставляют интерфейс Parquet. Для установки conda вы можете воспользоваться
conda install fsspec zarr pyarrow. Образ Docker tiagoantao/python-performance-dask
содержит все необходимые библиотеки. Давайте начнём с небольшого обзора библиотеки fsspec, которая позволит нам работать применяя один и тот же API с различными типами
файловых систем, причём как с локальными, так и с удалёнными.
Для хранения файлов существует множество систем, от почтенных локальных файловых систем, до облачных хранилищ, таких как Amazon S3, а также до подобных SFTP и SMB (совместные файловые ресурсы Windows) протоколов. Полный перечень большой, в особенности когда мы полагаем, что существует множество прочих подобных файловым системам объектов: к примеру, файл zip это контейнер файлов и каталогов, HTTP сервер обладает деревом обхода и так далее.
Работа с каждым типом файловой системы подразумевает изучение различных API программирования для каждой из них - трудоёмкая и даже вызывающая головную боль перспектива. Вникните в fsspec, библиотеку, которая создаёт уровень абстракции многих типов файловых систем позади унифицированного API. Пользуясь fsspec для взаимодействия со множеством типов файловых систем вам потребуется изучить лишь единственный API. При этом имеется ряд причуд: например, вы не можете ожидать что локальная файловая система будет вести себя ровно так же как удалённая, но эта библиотека существенно упрощает доступ к файловым системам при минимальных издержках.
Чтобы проиллюстрировать как работает fsspec, мы воспользуемся им для перемещения по репозиторию GitHub в поиске файлов zip с последующим определением содержат ли эти файлы zip CSV файлы. В данном упражнении мы рассматриваем GitHub как если бы он являлся файловой системой. Это не столь надуманно, как может показаться. Когда вы задумаетесь о нём, репозиторий GitHub это некое дерево каталога с упорядоченным по версиям содержимым.
В качестве образца репозитория мы воспользуемся репозиторием данной книги. В 08-persistence/01-fspec вы обнаружите файл zip
с именем dummy.zip, который содержит два CSV файла dummy. Наш код пройдётся по этому
репозиторию, отыщет файлы zip - в нашем случае имеется только dummy.zip - откроет его и воспользуется командой
describe pandas для обобщения всех CSV.
Давайте начнём с доступа к самому репозиторию при помощи fsspec и перечисления его корневого каталога:
from fsspec.implementations.github import GithubFileSystem
git_user = "tiagoantao"
git_repo = "python-performance"
fs = GithubFileSystem(git_user, git_repo)
print(fs.ls(""))
Мы импортировали класс GithubFileSystem, передали значения имён пользователя и репозитория и выдали список каталога
верхнего уровня. Обратите внимание, что значение корневого каталога представлено значением пустой строки, а не обычным значением
/. fsspec для доступа к хранилищу предоставляет большое число прочих классов, таких как локальная файловая система,
сжатые файлы, Amazon S3, HTTP, SFTP и тому подобные.
Полученный объект fs обладает рядом методов, распространённых в интерфейсах файловых систем Python. К примеру, для
обхода такой файловой системы, который нам требуется для поиска всех файлов zip имеется метод walk, который очень похож на
метод walk модуля os:
def get_zip_list(fs, root_path=""):
for root, dirs, fnames in fs.walk(root_path):
for fname in fnames:
if fname.endswith(".zip"):
yield f"{root}/{fname}"
get_zip_list это генератор, который вырабатывает все полные пути существующих файлов zip. Обратите внимание, что
данный код в точности тот, который мы применяли бы с os.walk, если бы root_path
был /.
Ограничения интерфейса fsspec
Хотя fsspec и предоставляет универсальный и простой интерфейс для файловых систем, он не способен скрывать все семантические отличия. Действительно, в некоторых случаях мы не хотим скрывать все имеющиеся отличия. В случае использования в качестве образца
GitHubFileSystem, здесь имеются две возможные ситуации, где могут наблюдаться отличия:Дополнительные функциональные возможности - вы можете перемещаться по данному репозиторию в любой момент времени, а не только в текущий момент времени его главной ветки. Вы способны указать ветку или тег и fsspec позволит вам проверить репозиторий именно в этом пункте.
Ограничения - не только у вас самих будут типичные проблемы с расположенными удалённо файловыми системами (скажем, если вы не подключены к Интернету, ваш код не будет работать), но и когда вы будете запрашивать большое число раз, он будет ограничивать вас по скорости.
Теперь, когда у нас имеется список zip в рассматриваемом репозитории, в качестве первого, естественного решения, мы скопируем эти файлы zip из данного репозитория в свою локальную файловую систему. Основная идея здесь состоит в том, что мы раскроем их локально чтобы посмотреть имеют ли они файлы CSV:
def get_zips(fs):
for zip_name in get_zips(fs):
fs.get_file(zip_name, "/tmp/dl.zip")
yield zip_name
Сейчас мы можем выполнить инспекцию своего файла внутри. Для этого мы можем, опять же наивно, воспользоваться встроенным
модулем zipfile Python:
import zipfile
import pandas as pd
def describe_all_csvs_in_zips(fs):
for zip_name in get_zips(fs):
my_zip = zipfile.ZipFile("/tmp/dl.zip") (1)
for zip_info in my_zip.infolist(): (2)
if not zip_info.filename.endswith(".csv"):
continue
print(zip_info.filename)
my_zip_open = zipfile.ZipFile("/tmp/dl.zip")
df = pd.read_csv(zipfile.Path(my_zip_open,
➥ zip_info.filename).open())
print(df.describe());
(1) Мы открываем данный файл применяя здесь модуль
zipfile.
(2) Обратите внимание, что модуль infolist
специфичен для модуля zipfile и с ним необходимо ознакомиться.
Обратите внимание на новый API, который нам требуется изучить для zipfile. Мы начали с его конструктора
и затем воспользовались методом infolist, однако нам может потребоваться повторно открыть промежуточный перечень zip по причине
семантики zipfile.
Наш предыдущий листинг кода это просто образец иллюстрации той путаницы, от которой спасает нас fsspec. fsspec предоставляет некий интерфейс для файлов zip, поэтому данный код мы можем переписать следующим образом:
from fsspec.implementations.zip import ZipFileSystem
def describe_all_csvs_in_zips(fs):
print(zip_name)
for zip_name in get_zips(fs):
my_zip = ZipFileSystem("/tmp/dl.zip")
for fname in my_zip.find(""): (1)
if not fname.endswith(".csv"):
continue
print(fname)
df = pd.read_csv(my_zip.open(fname)) (2)
print(df.describe())
(1) Этот метод find, как и все прочие,
имеется для всех видов файловых систем, не только для zip.
(2) Как и в случае с методом find,
open также доступен для всех видов файловых систем.
Помимо создания объекта ZipFileSystem, весь интерфейс в точности такой же как и у GitHub и очень близок к обычным
интерфейсам файлов Python. Нет необходимости в изучении интерфейса zipfile.
Вы можете применять fsspec для открытия файлов напрямую, хотя его семантика слегка отличается от стандартного open.
Например, чтобы открыть при помощи open fsspec файл zip, мы применяем такой код:
dlf = fsspec.open("/tmp/dl.zip")
with dlf as f: (1)
zipf = zipfile.ZipFile(f) (2)
print(zipf.infolist())
dlf.close()
(1) Для открытия файла нам требуется воспользоваться оператором
with.
(2) Для синтаксического разбора этого файла мы снова пользуемся модулем
zipfile Python.
Вот получаемый вывод:
[
<ZipInfo filename='dummy1.csv' filemode='-rw-rw-r--' file_size=22>,
<ZipInfo filename='dummy2.csv' compress_type=deflate
filemode='-rw-rw-r--' file_size=56 compress_size=54>
]
Обратите внимание на то, что для получения надлежащего дескриптора файла, нам требуется воспользоваться диалектом with
после open, что отличается от обычного подхода простого применения функции open.
Давайте вернёмся обратно в своему файлу zip внутри репозитория GitHub. Заметьте, что поскольку мы сможем рассматривать свой файл zip как контейнер для файлов,
такой файл подобен обладающей внутри себя другой файловой системой. fsspec обладает декларативным способом , который позволяет нам достаточно просто получать свои
данные: цепочками URL. Порой мы можете взять некий поток и воспринимать его как некую файловую систему.Проще рассмотреть это на примере; давайте выведем на печать
содержимое dummy1.csv:
d1f = fsspec.open("zip://dummy1.csv::/tmp/dl.zip", "rt")
with d1f as f:
print(f.read())
Обратите внимание на соединение в цепочку URL в действии: мы берём dummy1.csv из /tmp/dl.zip.
Вам нет нужды открывать этот файл; за вас об этом позаботится fsspec.
Помните как мы ссылались на свою реализацию get_zips как на наивную? Она
была наивной, поскольку нам на самом деле не пришлось в явном виде выгружать этот файл за счёт учтивости цепочки URL:
d1f = fsspec.open(
"zip://dummy1.csv::github://tiagoantao:python-performance@/08-"
"persistence/sec1-fsspec/dummy.zip")
with d1f as f:
print(pd.read_csv(f))
Для пояснения примера в явном виде мы жёстко кодируем полную цепочку URL.
Теперь, раз fsspec абстрагирован от взаимодействия с файловой системой, очень просто менять реализацию файловой системы. К примеру, давайте заменим GitHub на локальную файловую систему. Это так же просто как:
import os
from fsspec.implementations.local import LocalFileSystem
fs = LocalFileSystem()
os.chdir("../..")
Здесь предполагается, что вы исполняете данный сценарий в каталоге 08-persistence/sec1-fsspec
а раз так, ../.. будет корнем всего репозитория книги.
Вместо GitHubFileSystem мы применяем LocalFileSystem и это
почти то же самое. Поскольку мы исполняем данный код на два уровня глубже вершины своего репозитория, нам необходимо
вверх по дереву, chdir. Теперь наш код работает поверх локальной файловой системы, а не в GitHub. Например, выполните
describe_all_csvs_in_zips(fs).
Наконец, не лишним будет отметить, что PyArrow, о котором мы говорили в предыдущей главе, может напрямую взаимодействовать с fsspec:
from pyarrow import csv
from pyarrow.fs import PyFileSystem, FSSpecHandler
zfs = ZipFileSystem("/tmp/dl.zip")
arrow_fs = PyFileSystem(FSSpecHandler(zfs))
my_csv = csv.read_csv(arrow_fs.open_input_stream("dummy1.csv"))
Самый важный момент здесь заключается в том, что Arrow обладает понятием соответствующей файловой системы, что позволяет ему естественным образом интегрироваться
с fsspec. Файловая система Arrow способно выступать мостом с fsspec через pyarrow.fs.FSSpecHandler. После того как файловая
система fsspec поставлена таким образом в соответствие, можно прозрачно поверх неё пользоваться примитивами файловой системы Arrow.
![[Совет]](/common/images/admon/tip.png) | Совет |
|---|---|
|
fsspec поддерживает возможность частичной выгрузки данных с удалённых серверов, что может быть важным в ситуациях с Большими данными, когда нам требуется
лишь некий фрагмент большого файла. Это можно осуществлять только в тех случаях, когда соответствующий тип сервера, которым мы пытаемся воспользоваться,
поддерживает частичную выгрузку файла. К примеру, GitHub не поддерживает этого; и наоборот, S3 поддерживает. Вы можете включить такую функциональную
возможность активировав имеющееся кэширование при вызове |
Это был небольшой отскок, так как fsspec не имеет прямого отношения к производительности, хотя она и используется во многих связанных с производительностью библиотеках, таких как Dask, Zarr и Arrow. Теперь давайте вернёмся к нашему обычному программированию по расписанию и рассмотрим подходы к действенному хранению разнородных данных в виде столбцов, также именуемых кадрами данных.
Хранение данных в CSV сопряжено с проблемами. Во- первых, поскольку они не способны приспосабливать под тип каждый столбец, нередко в столбцах появляются неожиданные значения. Кроме того, сам их формат не эффективен. К примеру, вы могли бы гораздо компактнее представлять числа в двоичном представлении, нежели в текстовом виде. Кроме того, у вас нет возможности перехода к определённой строке или столбцу за постоянное время, так как невозможно вычислять их местоположение по той причине, что всякая строка в CSV может изменяться по размеру.
Для действенного хранения неоднородных данных в столбцах наиболее распространённым форматом стал Apache Parquet. Он подразумевает, что вы можете получать доступ всего к тому столбцу, который вам требуется и к тому же пользоваться сжатием данных и форматами кодирования столбцов для увеличения производительности.
В данном разделе мы изучим как применять Parquet для хранения кадров данных, опираясь на данные такси Нью- Йорка из своей предыдущей главы. По мере выполнения данной задачи я также представлю тур по многим функциональным возможностям Parquet.
![[Предостережение]](/common/images/admon/warning.png) | Предостережение |
|---|---|
|
Parquet это берущий своё начало в мире java формат файла, в особенности, в экосистеме Hadoop. Хотя имеющиеся в доступности реализации Python исключительно подходят под промышленные цели, они не реализуют установленную спецификацию полностью. Например, мы не можем определять во всех подробностях как бы мы хотели кодировать столбцы; также у нас нет возможности проверять как хранится столбец - кое- что я покажу здесь. Однако для подавляющего большинства вариантов применения необходимый функционал присутствует и со временем будет только увеличиваться. |
Просто напомним, что наш набор данных такси содержит сведения обо всех рейсах такси в Нью- Йорке за определённый период времени. Сведения включают, помимо прочего,
время начала и окончания поездки, место начала и окончания, стоимость, налоги и чаевые. Мы начинаем с того же самого файла что и в предыдущей главе, который содержит
рейсы такси за январь 2020 года. Первое, что мы сделаем, это преобразуем тот файл CSV в Parquet. Для этого мы воспользуемся Apache Arrow, представленный в нашей
предыдущей главе. Весь код можно найти в 08-persistence/.
import pyarrow as pa
from pyarrow import csv
import pyarrow.parquet as pq
table = csv.read_csv(
"../../07-pandas/sec1-intro/yellow_tripdata_2020-01.csv.gz")
pq.write_table(table, "202001.parquet")
Мы просто применяем write_table из своего модуля Parquet PyArrow. Заканчиваем мы двоичным файлом в 111 МБ. Сжатый CSV
составлял 105 МБ, а наша первоначальная версия без сжатия 567 МБ. Поскольку Parquet это структурированный двоичный формат, мы должны ожидать некоторые отличия в
размере для кое- какого содержимого. Основной момент тут состоит в том, чтобы не замыкаться на имеющихся деталях, но получить внутреннее представления о взаимосвязи
с имеющимся размером.
Давайте познакомимся с некоторыми функциональными возможностями Parquet через инспекцию своего файла:
parquet_file = pq.ParquetFile("202001.parquet")
metadata = parquet_file.metadata
print(metadata)
print(parquet_file.schema)
group = metadata.row_group(0)
print(group)
Вот сокращённый вывод:
<pyarrow._parquet.FileMetaData object at 0x7f90858879f0>
created_by: parquet-cpp-arrow version 4.0.0
num_columns: 18
num_rows: 6405008
num_row_groups: 1
format_version: 1.0
serialized_size: 4099
<pyarrow._parquet.ParquetSchema object at 0x7f9193aeed00>
required group field_id=0 schema {
optional int32 field_id=1 VendorID (Int(bitWidth=8, isSigned=false));
optional int64 field_id=2 tpep_pickup_datetime (
Timestamp(isAdjustedToUTC=false, timeUnit=milliseconds,
is_from_converted_type=false, force_set_converted_type=false));
....
<pyarrow._parquet.RowGroupMetaData object at 0x7f90858ad0e0>
num_columns: 18
num_rows: 6405008
total_byte_size: 170358087
Мы начали с вывода на печать имеющихся для нашего файла метаданных. Здесь мы получаем некоторые сведения, например о наличии 18 столбцов и 6 408 008 строк. Parquet также сообщает нам что в данном файле имеется единственная группа строк. Группа строк это раздел из всех строк: в больших файлах может иметься более одной группы строк. Группа строк будет содержать все данные столбца для всех строк в своей группе. Помните, что сведения в Parquet организованы по столбцам. Это вскорости станет очевидным.
Затем мы выведем на печать имеющуюся схему своего файла. Вот сокращённая версия:
required group field_id=0 schema {
optional int32 field_id=1 VendorID (Int(bitWidth=8, isSigned=false)); (1)
optional int64 field_id=2 tpep_pickup_datetime (
Timestamp(isAdjustedToUTC=false, timeUnit=milliseconds,
is_from_converted_type=false, force_set_converted_type=false));
optional double field_id=5 trip_distance;
optional binary field_id=7 store_and_fwd_flag (String);
}
(1) Здесь у нас имеется определение VendorID,
который обладает шириной в 8 бит без знака.
Данный код перечисляет все имеющиеся столбцы для своих данных. Например, VendorID это
int32, однако обратите внимание, что значение ширины бит это 8 и что они без знака. VendorID
имел лишь два возможных значения плюс null, а потому есть смысл уменьшить его реализацию лишь до 8 бит без знака. Теоретически, её можно даже снизить до ещё меньшего
числа бит, ибо Parquet поддерживает такое снижение.
Затем у нас имеется tpep_pickup_datetime, выступающий временной меткой. С точки зрения хранения данный элемент времени
это наиболее важная переменная, поскольку бо́льшая точность потребует больше пространства. pandas по умолчанию установлена на точности в наносекунду. Кроме
того, обратите внимание на store_and_fwd_flag: текст сохраняется в виде общих двоичных данных.
Давайте теперь взглянем на существующие метаданные для нескольких столбцов:
tip_col = group.column(13) # tip_amount
print(tip_col)
Вот сокращённый вывод:
physical_type: DOUBLE
num_values: 6405008
path_in_schema: tip_amount
statistics: (1)
has_min_max: True
min: -91.0
max: 1100.0
null_count: 0
distinct_count: 0
num_values: 6405008
physical_type: DOUBLE
logical_type: None
converted_type (legacy): NONE
compression: SNAPPY (2)
encodings: ('PLAIN_DICTIONARY', 'PLAIN', 'RLE')
has_dictionary_page: True
(1) Статистические сведения о данном столбце начинаются здесь.
(2) В этом столбце применяется данный алгоритм сжатия.
Наши метаданные начинаются со значения физического типа, общего числа значений и названия этой колонки. Имеющиеся статистические сведения (здесь, похоже, присутствует ошибка при вводе, отрицательное значение чаевых) показывают $-91 в качестве минимума, а $1 000 как максимум чаевых. Теперь всё становится действительно интересным.
По отношению хранения данных Parquet обладает способностью сжатия столбцов, что сохраняет место на диске. Сжатие столбцов также может обеспечивать потенциальную выгоду вычислений, связанную с задачами управления кэшем, которые обсуждались в нашей предыдущей главе. Разные столбцы могут обладать разными типами сжатия или вообще не обладать им.
В нашем образце применяется алгоритм Snappy. Snappy приносит в виде компромисса большее сжатие для скорости по сравнению, скажем, с gzip, который также рассматривается в качестве варианта. Обязательно проверьте какие методы сжатия реализует Arrow на момент применения. Для сравнительного анализа у Facebook имеются некоторые сведения.
Например, вы можете воспользоваться ZSTD:
pq.write_table(table, "202001_std.parquet", compression="ZSTD")
В данном образце мы применяем ZSTD для всех столбцов. В таком случае вы получите уменьшение со 110 МБ для Snappy до 82 МБ.
Parquet также способен кодировать столбцы не только непосредственными значениями, но также и пользоваться словарями, в которых длинные значения преобразуются в некую косвенную ссылку, что в потенциале сберегает гигантский объём пространства диска. Чтобы разобраться с тем как это помогает, предположим что чаевые представлены двойной точностью, требующей 64 бит, в то время как для чаевых имеется только 3626 различных значений:
print(len(table["tip_amount"].unique()))
Словарь способен снизить кодирование с 64 бит до 12 бит для значения, которых достаточно для кодирования до 4 096 значений. Нам также потребуется словарь для
хранения, который выступает остатком для 3 626 значений. Тем не менее, поскольку у нас имеется большое число различных значений, это может превращать применение
словаря в несущественное. При помощи write_table вы можете контролировать будет ли столбец храниться со словарём или нет.
И последнее, но не в отношении значимости, обратите внимание на то, что наше кодирование также обладает RLE, которое означает сокращение для Run Length Encoding
(кодирование длины последовательности). Давайте взглянем на преимущество RLE на слегка глупом примере. Давайте создадим кадр данных для столбца с
VendorID, за которым следует ещё один столбец тоже с
VendorID, но в таком порядке:
import pyarrow.compute as pc
silly_table = pa.Table.from_arrays([
table["VendorID"],
table["VendorID"].take(
pc.sort_indices(table[""endorID"]))],
["unordered", "ordered"]
)
Итак, это те же самые данные, причём с упорядоченными и не упорядоченными версиями. Давайте теперь посмотрим как много пространства каждый из этих столбцов занимает в файле Parquet:
pq.write_table(silly_table, "silly.parquet")
silly = pq.ParquetFile("silly.parquet")
silly_group = silly.metadata.row_group(0)
print(silly_group.column(0))
print(silly_group.column(1))
Наш неупорядоченный файл требует 953 295 байт, а для упорядоченного файла требуется 141 байт! Тот способ, коим работает RLE, состоит в сохранении самого значения
и числа его повторов. Для упорядоченного столбца VendorID у нас имеется экстремальная ситуация: у нас есть только три
значения (1, 2 и null), причём они упорядочены.
Поэтому, теоретически, RLE способен хранить: 1.0 2094439 / 2.0 4245128 / null 65441.
RLE способен сжимать данные достаточно существенно. Хотя нашем случай экстремален в плане эффективности, обычно RLE хорошо работает для упорядоченных полей или полей небольшим числом значений. Тем не менее, когда вы отклоняетесь от этих предположений, обязательно оцените получаемые вами преимущества сжатия.
Файлы меньшего размера помогают в хранении и времени обработки. Вспомните по Главе 6, что если вы можете хранить данные в более быстрых типах памяти, порой вы можете увеличивать производительность на порядки.
Данный формат является расширяемым, а потому со временем можно ожидать разработки нового способа действенного хранения данных. Формат также допускает разделение данных на разделы, что имеет ряд преимуществ с точки зрения эффективности. Давайте проясним это на примере.
Чтобы пояснить что собой представляет разбиение на разделы и что влечёт за собой данный процесс, давайте разобьём на разделы свой набор данных, воспользовавшись
VendorID и passenger_count. Поскольку разделы не могут основываться на значениях
null, мы удалим их из своего набора данных. Мы делаем это только в качестве примера; обычно вы не можете удалять строки со значениями null просто для удобства:
tip_col = group.column(13) # tip_amount
print(tip_col)
Вот сокращённый вывод:
from pyarrow import csv
import pyarrow.compute as pc
import pyarrow.parquet as pq
table = csv.read_csv(
"../../07-pandas/sec1-intro/yellow_tripdata_2020-01.csv.gz")
table = table.filter(
pc.invert(table["VendorID"].is_null())) (1)
table = table.filter(pc.invert(table["passenger_count"].is_null()))
pq.write_to_dataset(
table, root_path="all.parquet",
partition_cols=["VendorID", "passenger_count"])
(1) Снова обращаем ваше внимание на то, что синтаксис проведения вычисления при помощи Arrow сильно отличается от pandas.
Эквивалентом для нашей самой первой строки фильтрации был бы table = table[~table["VendorID"].isna()].
Если вы взглянете на all.parquet, вы обнаружите пару сюрпризов: Самый большой из них состоит в том, что в каталоге
больше нет файла! Наше сокращённое содержимое выглядит как-то так:
.
├── VendorID=1
│ ├── passenger_count=0
│ │ └── e59ac47b5193411e9772bfee9d423d61.parquet
│ ├── passenger_count=1
│ │ └── ee90fe5b818d4a37a32b5a415915610b.parquet
│ └── passenger_count=9
│ └── 002ff0bba1d340abb6174c5c64f779d7.parquet
└── VendorID=2
├── passenger_count=0
│ └── 5809e29649524202a9b3cef5371c46d9.parquet
└── passenger_count=9
└── feaff7a23bbf4ae2b687b34dcaa10afb.parquet
Данная структура каталога отражает нашу стратегию разбиения на разделы. Самый первый уровень каталогов обладает записью для
VendorID, а второй записью для passenger_count.
Теперь у вас есть два варианта. Самый простой - и обоснованно наименее интересный - загружать всю таблицу:
all_data = pq.read_table("all.parquet/")
Так вы получите все свои данные как обычную таблицу. В качестве альтернативы, вы можете с тем же эффектом сделать следующее:
dataset = pq.ParquetDataset("all.parquet/")
ds_all_data = dataset.read()
Однако, как иной вариант, вы также способны загружать каждый файл parquet по отдельности. Например, давайте загрузим свой файл для раздела с идентификатором поставщика 1 и с тремя пассажирами:
import os
data_dir = "all.parquet/VendorID=1/passenger_count=3"
parquet_fname = os.listdir(data_dir)[0] (1)
v1p3 = pq.read_table(f"{data_dir}/{parquet_fname}")
print(v1p3)
(1) Название нашего файла parquet не обеспечено, о потому мы получаем самый первый файл.
Если вы взглянете на получаемый вывод, вы обнаружите, что наши столбцы VendorID и
passenger_count пропущены, поскольку они подразумеваются по своему каталогу.
![[Предостережение]](/common/images/admon/warning.png) | Предостережение |
|---|---|
|
Что именно расположено в каталоге может разниться. В нашем случае, с применением PyArrow, это единственный файл Parquet. Например, вы можете сообщить Parquet далее расщеплять каждый раздел на файлы по группам строк. Таким образом, убедитесь что вы изучили как в действительности записаны данные на диск и приспосабливайте код соответствующим образом. |
В чём смысл разбиения на разделы с точки зрения производительности? Теперь мы имеем возможность загружать каждый файл Parquet по- отдельности и соответствующим образом обрабатывать каждый из них. Например, мы можем повысить производительность, применяя несколько процессоров в одном компьютере, причём каждый из них выполняет анализ для каждого из файлов. Мы даже можем обрабатывать разные файлы в разных машинах. Неявно такая файловая система может быть более действенной, поскольку параллельная загрузка осуществляется с разных частей диска. Также может иметься выигрыш в оперативной памяти, потому как мы не не загружаем столбцы разбиения на разделы. Наконец, разбиение на разделы открывает магистральный путь для параллельной записи, что обеспечивает прирост производительности за счёт параллельности. Более подробно одновременную запись мы обсудим в Разделе 8.4.
Тот способ, коим данные разбиваются на разделы существенен с точки зрения производительности. Например, Vendor 1 обладает половиной данных Vendor 2, что означает,
что стоимость обработки Vendor 2, вероятно, будет удвоена по сравнению с Vendor 1. Такое удвоение может вызвать для вас ожидание самого медленного из всех разделов,
потому как вы хотите быть как можно более универсальным. VendorID может оказаться хорошим выбором по сравнению с
passenger_count. Parquet обладает намного бо́льшей функциональностью, однако с точки зрения производительности у нас
имеется хороший обзор того, как мы можем извлекать из данного формата пользу.
В данном разделе мы будем работать с файлами Parquet и CSV чтобы рассмотреть два простых метода работы с данными, имеющими размер больше оперативной памяти: отображение памяти и фрагментация. Существуют более изощрённые способы осуществления обеих задач, и мы обсудим их в разделе 8.4, а также в своей следующей главе. Тем не менее, фрагментация и отображение памяти это важные понятия, ложащиеся в основу более сложных библиотек. По этой причине разобраться с ними важно не только само по себе, но и имеет основополагающее значение для понимания более современных методов.
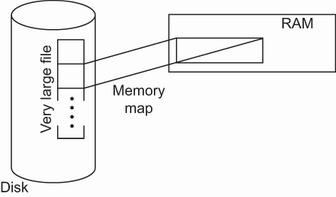
Отображение памяти происходит когда часть памяти напрямую приводится в соответствие части файловой системы. В конкретном случае с NumPy, над постоянно содержащемся в хранилище массивом можно производить оценки при помощи обычного API NumPy и о переносе в оперативную память любых требующихся нам частей такого массива позаботится NumPy. В большинстве ситуаций это осуществляется прозрачно ядром операционной системы для NumPy. И наоборот, это изменяет постоянно хранимое представление при осуществлении нами записи. Поскольку оценки выполняются в оперативной памяти, это может ускорять ваш код на несколько порядков. Рисунок 8.1 показывает отображение памяти.
В данном случае мы будем пользоваться простым абстрактным примером создания большого массива и доступа к нему. Для данного упражнения я рекомендую размер больше вашей оперативной памяти, однако для которого у вас достаточно дискового пространства. Выделение выполняется достаточно просто:
import numpy as np
SIZE_IN_GB = 10 (1)
array = np.memmap("d\"ata.np", mode="w+",
dtype=np.int8, shape=(SIZE_IN_GB * 1024, 1024, 1024))
print(array[-1, -1, :10])
print(v1p3)
(1) Измените этот размер на подходящее вашей машине, как это описано ранее, значение.
Вызов np.memmap достаточно прямолинеен: вы передаёте имя файла, режим открытия и значение и типа с контуром (shape) данного
массива. Если вы выдадите перечень файлов на своём диске, вы обнаружите файл с размером в 10 ГБ.
Данный массив будет инициирован всеми нулями; следовательно, наш вывод выдаст на печать некий массив из 10 нулей. Добавим теперь 2 ко всем элементам этого массива:
array += 2
Его интерфейс в точности такой же как для размещаемого в памяти массива NumPy, однако вы заметите, что данная операция займёт несколько секунд. Значение времени выросло потому как наш большой файл изменяется повсеместно; это не быстрая операция в оперативной памяти.
Давайте теперь откроем этот файл и выведем на печать его последнее значение:
array = np.memmap("data.np", mode="r",
dtype=np.int8)
print(array.shape)
print(array[:-10])
Вот вывод:
(10737418240,)
[2 2 2 ... 2 2 2]
Здесь важным моментом является то, что контур массива не хранится вместе с ним, поэтому, если вы его отобразите без определения значения формы, вы получите линейный массив. Поэтому вам требуется убедится, что вы восстанавливаете желаемый контур. После этого мы напечатаем последние 10 элементов данного массива и получим десять двоек.
![[Замечание]](/common/images/admon/note.png) | Копирование записью NumPy |
|---|---|
|
Отображение памяти NumPy позволяет нам применять методику, имеющую название копирования записью (copy- on- write). Это делает для вас возможным обладать загруженными в память несколькими копиями дискового массива и платить за использование памяти значительно меньше. Данная методика во многих ситуациях подвержена ошибкам, в основном потому, что Python не лучший язык для работы с совместно используемыми структурами данных, а также по той причине, что семантика отображения памяти становится запутанной при внесении изменений в базовый файл. Я не думаю, что получаемые преимущества оправдывают имеющиеся риски, если только вы не уверены, что будете выполнять только операции считывания. Если вы хотите ознакомиться с данной методикой, я рекомендую прекрасную статью Итамара Тернера-Трауринга. В обычной ситуации я бы избегал явных методов отображения памяти, которые осуществляют одновременную запись при совместном использовании, только если вы не абсолютно уверены, что все процессы выполняют только считывание. Кроме того, если вы являетесь разработчиком библиотеки нижнего уровня, вы можете пользоваться отображением памяти пр записи, но, скорее всего, вы всё равно не будете применять Python для реализации наиболее действенных частей кода, а потому данная задача будет решаться иными языками программирования. |
Помните, что даже когда вы не пользуетесь отображением памяти в явном виде, многие применяемые вами инфраструктуры будут осуществлять это косвенным образом, а потому в этом важно разбираться. Давайте теперь обсудим другую методику работы с большими файлами: фрагментацию.
Фрагментация, как и предполагает её название, означает обработку файла порциями (chunks, пробелами). Вы считываете (или выполняете запись) своего файла по частям. Вы определённо будете работать с фрагментацией когда будете применять Zarr (см. радел 8.4) или Dask (см. главу 10).
Здесь мы вернёмся к своему проверенному примеру такси. Мы будем выполнять преобразование своего файла из CSV в Parquet, но на этот раз фрагментами. Хотя этот наш файл и достаточно мал чтобы мы имели возможность осуществления этого в оперативной памяти большинства компьютеров, давайте предположим что мы в ограниченной по памяти машине и что загрузка данного файла целиком в оперативную память невозможна.
Для считывания своего файла CSV мы воспользуемся pandas, а для записи версии Parquet применим Arrow. Мы можем всё это выполнять в Arrow, что было бы даже более эффективно, но мы желаем продемонстрировать интерфейс фрагментации pandas:
import pandas as pd
table_chunks = pd.read_csv(
"../../07-pandas/sec1-intro/yellow_tripdata_2020-01.csv.gz",
chunksize=1000000
)
print(type(table_chunks)) (1)
for chunk in table_chunks: (2)
print(chunk.shape)
(1) Значением типа будет pandas.io.parsers.TextFileReader.
(2) Каждый фрагмент будет кадром данных.
Нам только потребуется добавить свой параметр chunksize для read_csv. У вас не будет
кадра данных из read_csv, а вместо него генератор фрагментов. Каждый фрагмент затем будет кадром данных с максимальным
размером в 1 миллион строк.
Теперь мы выполняем надлежащим образом преобразование. Самый первый необходимый для осуществления нами момент состоит в повторном открытии данного файла. Нам придётся по разу выполнить итерацию по всем имеющимся фрагментам, а потому нам придётся вернуться обратно в самое начало:
table_chunks = pd.read_csv(
"../../07-pandas/sec1-intro/yellow_tripdata_2020-01.csv.gz",
chunksize=1000000,
dtype={
"VendorID": float,
"passenger_count": float,
"RatecodeID": float,
"PULocationID": float,
"DOLocationID": float,
"payment_type": float,
}
)
Нам также требуется задать типы данных некоторых столбцов; значения типов некоторых столбцов будут изменяться от фрагмента к фрагменту. В основном это имеет
место для обладающих значением null столбцов. Когда имеются значения null, значение типа будет повышаться до float,
поскольку в pandas не существует способа представления null целыми.
Теперь мы пройдёмся по своим фрагментам и создадим файл Parquet:
first = True
writer = None
for chunk in table_chunks:
chunk_table = pa.Table.from_pandas(chunk) (1)
schema = chunk_table.schema
if first:
first = False
writer = pq.ParquetWriter(
"output.parquet", schema=schema) (2)
writer.write_table(chunk_table)
writer.close()
(1) Мы выполняем преобразование кадра pandas в таблицу Arrow.
(2) Мы создаём объект писателя. При инициализации нам потребуется задать значение схемы.
Применяемый интерфейс ParquetWriter позволяет нам выполнять запись таблицы за таблицей в одном и том же файле.
Каждая таблица будет записана в обособленной группе строк Parquet. По существу, это будет некий фрагмент.
Считать данные parquet мы можем различными способами:
pf = pq.ParquetFile("output.parquet")
print(pf.metadata)
for groupi in range(pf.num_row_groups): (1)
group = pf.read_row_group(groupi)
print(type(group), len(group))
break
table = pf.read()
table = pq.read_table("output.parquet")
(1) Мы способны считывать каждую группу строк по отдельности.
Получаемые метаданные нашего файла Parquet будут указывать что имеется семь групп строк. Parquet позволяет нам считывать группу строк за группой строк. Если
у вас имеется достаточно оперативной памяти, существует два интерфейса - в ParquetFile при помощи
read или в модуле parquet с применением read_table
- которые заботятся о считывании всех групп чтения и создании таблицы в оперативной памяти.
Вооружившись пониманием фрагментации, которая позволяет нам загружать и обрабатывать свои данные по частям, теперь мы намерены взглянуть на Zarr. Zarr это библиотека, позволяющая нам манипулировать очень большими однородными N- мерными массивами (то есть массивами NumPy).
Некоторые из самых больших из имеющихся наборов данных представляют собой не неоднородные кадры таблиц данных, а многомерные однородные массивы. Таким образом, важно выполнять действенное хранение таких больших массивов.
Zarr предоставляет нам возможность эффективного хранения однородных многомерных массивов при различных серверных основаниях и разнообразных форматах кодирования. Подобная параллельной записи функциональная возможность может оказываться чрезвычайно востребованной для действенного производства данных.
Для представления данных массива существует некое число весьма зрелых стандартов (например, NetCFD и HDF5), но в нашем случае мы будем применять зарождающийся формат Zarr. По сравнению с прочими форматами, Zarr значительно более оптимизирован под действенную обработку. Например, он допускает параллельную запись и различную организацию структуры файла, что может оказывать массированное воздействие на производительность. Параллельная запись делает возможной для большого числа одновременных процессов в одно и то же время работать с одной и той же структурой. Отличающиеся структуры файла позволяют нам пользоваться имеющимися свойствами производительности установленной файловой системы.
Хотя Zarr это файловый формат, он стартовал с реализованного пространства Python в библиотеке с названием Zarr. Поэтому вы можете быть уверенным, что соответствующая версия Python реализует все основные функциональные возможности данного формата. Если вы планируете применять Zarr в других языках программирования, вам следует сначала убедиться что имеющиеся для данного языка программирования библиотеки поддерживают подобные функциональные возможности. В определённом смысле Zarr выступает противоположностью Parquet: Parquet пришёл в Python из экосистемы Java, а потому Python для Parquet по- прежнему не покрывает все его функциональные возможности. Для Zarr же, именно Pytgon выступает золотым стандартом.
Zarr стартовал в пространстве биоинформатики и мы воспользуемся примером из биоинформатики. Мы будем применять данные из старого проекта генома с названием HapMap. Данный проект обладает вариантами геномов (отличающихся символами ДНК) для многих персонажей по популяции людей. Для данного упражнения вам нет нужды знакомиться с научными подробностями. Мы сообщим минимум понятий, необходимых нам для продолжения работы.
Например, мы начнём с предварительно подготовленной базы данных Zarr, которую я сгенерировал из данных HapMap в формате
Plink. Вам не стоит беспокоиться о первоначальном формате, однако если это вам
интересно, вы можете найти тот код, которым я сгенерировал базу данных Zarr и которым вы можете пользоваться в моём репозитории
в каталоге 08-persistence/sec4-zarr/hapmap. Предварительно подготовленный файл Zarr можно найти в
https://tiago.org/db.zarr.tar.gz. Он содержит генетические сведения 210 персоналий из
различных людских популяций.
Одной из наших целей будет создание ещё одной базы данных Zarr, которую можно будет применять для выполнения анализа основных компонентов (PCA, principal components analysis) - метода неконтролируемого машинного обучения - получившего распространение в науке о геноме - который потребует переформатирования имеющихся у нас в исходной базе данных сведений. Здесь мы не будем запускать PCA, а лишь подготовим до него необходимый файл.
Давайте начнём рассматривать что находится внутри нашей базы данных. По мере прохождения этой базы данных мы будем напоминать себе о необходимых задействованных геномных понятиях:
(1) Выводит на печать имеющуюся структуру содержимого исследуемого файла.
Zarr это дерево контейнеров для массивов, а потому у нас имеется структура каталога, в которой узлы листьев являются массивами. Вот усечённая версия нашего файла:
├── chromosome-1
│ ├── alleles (318558,) <U2
│ ├── calls (318558, 210) uint8
│ └── positions (318558,) int64
├── chromosome-10
│ ├──alleles (216535,) <U2
│ ├──calls (216535, 210) uint8
│ └── positions (216535,) int64
Данные расщепляются на хромосомы и для хромосомы существует иерархия. Каждая хромосома обладает перечнем позиций генотипов (для которых мы получаем буквы ДНК)
в positions. Все возможные аллели (то есть буквы ДНК) для позиции находятся в соответствующем массиве
alleles. Основная матрица находится в calls, в которой для имеющихся 210 отдельных
представителей у нас есть необходимые аллели для каждого маркера. Итак, в хромосоме 1 имеется 318 558 маркеров, причём размер матрицы
calls составит 318 558 x 210. Для всякого индивидуального представителя и каждого маркера имеются два вызова, которые
будут закодированы отдельным числом.
Наша цель заключается в создании составной матрицы всех вызовов для отправки в реализацию PCA. Не стоит беспокоиться о генетике; с нашей точки зрения важно то,
что мы имеем двумерную матрицу вызовов со значениями 0/1/2, кодируемыми целым числом без знака с 8 битами и два одномерных массива, причём один с 64- битными целыми
числами (positions), а другой со строками размером до двух символов (alleles).
Прежде чем мы углубимся в связанные с производительностью задачи, давайте вкратце обсудим как проходить по данным Zarr. Всю имеющуюся структуру мы можем обойти следующим образом:
def traverse_hierarchy(group, location=""):
for name, array in group.arrays(): (1)
print(f"{location}/{name} {array.shape} {array.dtype}")
for name, group in group.groups(): (2)
my_root = f"{location}/{name}"
print(my_root + "/")
traverse_hierarchy(group, my_root)
traverse_hierarchy(genomes)
(1) Выводит на печать имеющуюся структуру содержимого исследуемого файла.
(2) Выводит на печать имеющуюся структуру содержимого исследуемого файла.
Когда Zarr считывает файл, он возвращает объект Groupю Соответствующий метод
groups будет возвращать генератор со всеми подчинёнными группами внутри, а потому мы можем полагаться на него для обхода
репозитория Zarr.
Для доступа к содержимому вы также может воспользоваться простой подобной каталогу номенклатурой, которая зависит от вашего субъективного предпочтения. Например:
in_chr_2 = genomes["chromosome-2"]
pos_chr_2 = genomes["chromosome-2/positions"]
calls_chr_2 = genomes["chromosome-2/calls"]
alleles_chr_2 = genomes["chromosome-2/alleles"]
in_chr_2 будет обладать Group для ключей pos_chr_2,
calls_chr_2, а alleles_chr_2 будет иметь готовыми к применению соответствующие массивы
chromosome-2/positions, chromosome-2/calls и
chromosome-2/alleles.
Давайте получим из своей структуры некие сведения:
print(in_chr_2.info)
Вот предоставляемый вывод:
Name : /chromosome-2
Type : zarr.hierarchy.Group
Read-only : False
Store type : zarr.storage.DirectoryStore
No. members : 3
No. arrays : 3
No. groups : 0
Arrays : alleles, calls, positions
То что у нас имеется, это Group, содержащий трёх участников, причём все они оказываются массивами; внутри значения
имени могут присутствовать подчинённые группы.
Zarr поддерживает большое число хранимых типов: в нашем случае мы пользуемся zarr.storage.DirectoryStore, однако вы
можете обнаружить классы для применения в оперативной памяти, файлы zip, файлы DBM, SQL, fsspec и тому подобное.
Как мы вскорости обнаружим, DirectoryStore очень полезен при поддержке расширенных функциональных возможностей
одновременности, однако на текущий момент давайте взглянем на применяемую структуру данных. На тот случай, если вы не обратили на это внимание,
db.zarr это не файл, а каталог. Приводимый ниже код это усечённая версия структуры его каталога:
.
├── chromosome-1
│ ├── alleles
│ └── calls
│ └── positions
├── chromosome-10
│ ├── alleles
│ └── calls
│ └── positions
...
Эта структура каталога подражает структуре группы Zarr, что превращает разработку в более простую.
Давайте теперь обсудим как хранятся массивы, существенно наиболее сложный и занимательный предмет:
print(pos_chr_2.info)
Теперь мы организуем свой вывод несколько иначе и расщепим его на несколько частей. Давайте начнём с неких базовых сведений:
Type : zarr.core.Array
Data type : int64
Shape : (333056,)
Order : C
Read-only : False
Store type : zarr.storage.DirectoryStore
При помощи того что мы изучали в предыдущих главах вы должны быть способны интерпретировать эти сведения: нашим объектом выступает
zarr.core.Array с типом данных 64- битных целых и обладающий 335 056 числами, к тому же этот массив C- упорядочен и
в него разрешена запись.
Давайте взглянем на профиль фрагмента:
Chunk shape : (41632,)
Chunks initialized : 8/8
Напомним, что фрагментация это некий способ разбиения большого массива на разделы меньшими равными частями (фрагментами, chunks), которыми проще манипулировать. (Рисунок 8.2):
Рисунок 8.2
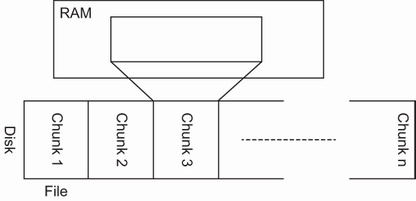
Файл большого массива может быть разбит на разделы фрагментами равного размера для обработки по- раздельности
Zarr сообщает нам, что каждый фрагмент обладает фрагментами с размером в 41 632 элемента; тем самым мы приходим к восьми фрагментам с 333 056 элементами. Когда мы создавали этот массив в своём сценарии поддержки создания предварительно подготавливаемой версии мы были слегка наивными и не указали значение размера фрагмента, а раз так, Zarr попытался догадаться о подходящем размере. Размер фрагмента может - и должен - определяться при создании. Позднее в этом разделе мы увидим почему.
Также обратите внимание, что все фрагменты инициализированы (то есть массив пустой). Фрагменты без инициализации потенциально способны сберегать большой объём дискового пространства. И опять же, мы обнаружим это позднее при создании массива.
Если вы пройдёте в каталог db.zarr/chromosome-2/positions, вы обнаружите восемь
файлов с именами от 0 до 7; именно они представляют по файлу на фрагмент. Такое
разделение при помощи Zarr упрощает параллельную запись - мудрёную функциональную возможность, которую не отыскать во многих системах хранения массивов.
Наконец, массивы Zarr могут сживаться, тем самым сохраняя большой объём дискового пространства, а потенциально и времени обработки, что обсуждалось ранее в данной главе. Вот часть общего вывода, описывающего это:
Compressor : Blosc(cname='lz4', clevel=5,
shuffle=SHUFFLE, blocksize=0)
No. bytes : 2664448 (2.5M)
No. bytes stored : 687723 (671.6K)
Storage ratio : 3.9
В нашем случае данные были сохранены с применением Blosc при помощи алгоритма LZ4. Первоначальный размер 2 664 448 байт - 333 056 элементов, помноженные на 8 байт для 64- битных целых с окончательным размером в 687 723 байта, тем самым сжимая в 3.9 раз. Принимая во внимание что такие массивы обладают однородным типом, следует ожидать, что в среднем сжатие превзойдёт общее сжатие разнородных кадров данных. Конечно, это ожидание для среднего случая; например, массив со случайными значениями очень сложно сжимать.
Для своего массива calls мы обладаем аналогичным выводом, тем не менее, приспособленным под два измерения. Вот обрезанная
версия вывода на печать print(calls_chr_2.info):
Shape : (333056, 210)
Chunk shape : (41632, 27)
Chunks initialized : 64/64
в данной ситуации у нас имеется матрица с размерами 333 056 x 210 и двумерными фрагментами.
![[Совет]](/common/images/admon/tip.png) | Совет |
|---|---|
|
Вы можете фрагментировать N- мерный массив размерностями меньшими N. Например, свой двумерный массив мы можем фрагментировать только одномерными. Такой выбор может иметь смысл когда вам необходимо обрабатывать в одно и то же время все сведения по одному измерению. Как и со всеми решениями о фрагментации, это определяется вашим конкретным случаем. |
Каждое измерение разделяется на восемь интервалов в общей сложности для 64 фрагментов. Если вы выведете перечень содержимого
db.zarr/chromosome-2/calls, вы обнаружите 64 файла, удобно именуемые X.Y, где X и Y изменяются от 0 до 7 и ссылается на
значение номера фрагмента по каждому из измерений.
Наконец, у нас имеется массив аллелей, которые представлены строками из двух символов (например, AT, CG, TC и т.д.). Вот усечённый вывод
print(alleles_chr_2.info):
Data type : <U2
Данный вывод это строка Unicode с фиксированным размеров в два байта. Напоминаем вам по Главе 2, что представление строк в Python изощрённое - или обременительное, в зависимости от избранной точки зрения - а оценка размера строки Python в байтах достаточно не тривиальна.
Для эффективности доступа полезно когда у нас присутствуют строки фиксированного размера и предсказуемый размер представления. Zarr предоставляет два встроенных представления строк: когда у вас имеются только символы ASCII, вы можете пользоваться массивом байт, а если у вас имеются символы помимо ASCII, Zarr обеспечивает представление Unicode фиксированного размера, в отличие от реализации строк Python переменной длины. Если вам требуются строки переменной длины с различным кодированием, Zarr также предоставляет для этого средства кодирования, однако будьте аккуратны с воздействием на производительность столь могучей гибкости; когда это возможно в отношении хранения, выделяйте строки с фиксированной длиной.
Теперь, когда у нас имеется обзор того как организованы данные Zarr, давайте создадим массив, который является составным из всех позиций по всем хромосомам. Поскольку PCA в качестве входных данных требуется единая матрица, нам нужна отдельная матрица из всех хромосом.
Теперь мы намерены создать новый массив, который может применяться для алгоритмов автоматического обучения, например,PCA. Это просто соединение всех массивов (то есть вызовов для всех хромосом).
Прежде чем мы начнём, нам требуется узнать размер того массива, который нам необходимо выделить. Для его определения мы пройдёмся по имеющемуся файлу Zarr для выделения значения числа маркеров хромосом, которое будет отличаться в зависимости от конкретной задачи:
import zarr
genomes = zarr.open("db.zarr")
chrom_sizes = []
for chrom in range(1, 23):
chrom_pos_array = genomes[f"chromosome-{chrom}/positions"]
chrom_sizes.append(chrom_pos_array.shape[0])
total_size = sum(chrom_sizes)
Этот код просто проверяет первое измерение всех одномерных массивов для позиций. Обладая этими сведениями мы можем вычислять значение размера всего массива Zarr без сжатия.
Поучив на руки значение общего размера мы можем выполнить выделение такого массива:
CHUNK_SIZE = 20000
all_calls = zarr.open(
"all_calls.zarr", "w",
shape=(total_size, 210), (1)
dtype=np.uint8, # type change
chunks=(CHUNK_SIZE,))
(1) 210 это число персональных представителей в нашем наборе данных.
Самый важный параметр в плане производительности это размер фрагмента. Мы выбираем значение, которое позволит нам превысить 1 МБ для фрагмента, хотя вам придётся для вашего конкретного случая отрегулировать размер фрагмента. В общей сложности 20 000 x 210 это около 4 МБ, однако мы выполняем подсчёт с неким сжатием. Мы полагаем, что все представители будут считаны одновременно, поэтому мы выполняем разбиение только по одному измерению. Не ограничивайте себя в изменении значения размера фрагмента, поскольку вы обнаружите чёткую разницу в производительности.
![[Замечание]](/common/images/admon/note.png) | Общие соображения для принятия решения размера фрагмента |
|---|---|
|
Очень сложно придумать общие правила для определения размера фрагмента. Вам потребуется рассмотреть свои алгоритмы и варианты применения. Между тем, вот основные правила:
|
Давайте получим сведения для all_calls. Вот усечённая версия:
Type : zarr.core.Array
Data type : uint8
Shape : (3976554, 210)
Chunk shape : (20000, 210)
No. bytes : 835076340 (796.4M)
No. bytes stored : 345
Storage ratio : 2420511.1
Chunks initialized : 0/199
Следует обратить внимание на то, что самая важная проблема состоит в значении числа хранимых байт и, соответственно, количество инициируемых фрагментов. Хотя
общий ожидаемый размер составляет 796.4 МБ, используется лишь 354 байт (!), потому как не были сохранены никакие данные (то есть не инициализирован ни один фрагмент).
По умолчанию, Zarr предполагает, что все значения в его массиве равны 0, когда они не инициированы. Если вы на данном этапе отобразите свой каталог
all_calls.zarr, вы обнаружите что он пуст и вообще не потребляет места.
На самом деле, скрытый файл с названием .zarray обладает некими метаданными. Если вы откроете этот файл, вы обнаружите
файл с версией JSON тех параметров, которые мы передали созданию массива Zarr помимо прочих параметров по умолчанию.
Теперь давайте создадим отдельный объединённый массив. Для анализа PCA нам требуется единый массив данных.
Мы обсудим две версии: первая это последовательная версия, а вторая одновременная. Вот первая:
def do_serial():
curr_pos = 0
for chrom in range(1, 23):
chrom_calls_array = genomes[f"chromosome-{chrom}/calls"]
my_size = chrom_calls_array.shape[0]
all_calls[curr_pos: curr_pos + my_size, :] = chrom_calls_array
curr_pos += my_size
do_serial()
print(all_calls.info)
Этот код просто последовательно копирует все вызовы хромосом для своего массива all_calls. Обратите внимание на то,
что всё управление хранением полностью абстрагировано поверх типичного интерфейса NumPy.
После выполнения этого кода, если мы выведем на печать сведения для all_calls, произойдёт несколько изменений:
No. bytes : 835076340 (796.4M)
No. bytes stored : 297035153 (283.3M)
Storage ratio : 2.8
Chunks initialized : 199/199
Теперь, когда все фрагменты инициированы, в хранилище занято 283.3 МБ - в соотношении хранения 2.8 по сравнению с 796.4 МБ для общего числа байт. Если вы
выведете перечень своего каталога all_calls.zarr, вы обнаружите 199 файлов: по одному для каждого фрагмента.
Весь предыдущий код требует для исполнения несколько секунд. Хотя я бы не стал просить вас запустить пример с большим числом терабайт данных, который потребует часов, просто заметить, что чем больше данных, тем значение для подобного преобразования может становиться запретительно длинным.
Итак, в качестве второй версии мы создадим одновременную версию, которая будет выполнять считывание из массивов хромосом и записывать в свой массив
all_calls. И считывание, и запись
будут одновременными.
Не так много библиотек поддерживает одновременную запись, но Zarr это делает. Помещая каждый фрагмент в обособленный файл, его каталог хранения упрощает реализацию одновременной запись. В подобной ситуации, простая архитектура основывается на свойствах производительности файловой системы и открывает возможность для очень важной функциональной возможности.
Теоретически можно выполнять запись любого предпочтительного для вас размера, но её осуществления в фрагмент за фрагментом будет наиболее действенным, поскольку Zarr не придётся иметь дело с параллельной записью в один и тот же файл. Фундаментальным моментом является то, что размер вашего фрагмента обязан подгоняться под ваш вариант применения и, если это допустимо, вам следует пробовать обрабатывать данные по частям.
В нашей ситуации, мы не можем перейти к упрощению обработки по хромосомам; нам придётся выполнять запись по фрагментам. Вот функция записи фрагмента целиком:
def process_chunk(genomes, all_calls, chrom_sizes, chunk_size, my_chunk):
all_start = my_chunk * chunk_size (1)
remaining = all_start
chrom = 0
chrom_start = 0
for chrom_size in chrom_sizes: (2)
chrom += 1
remaining -= chrom_size
if remaining <= 0:
chrom_start = chrom_size + remaining
remaining = -remaining
break
while remaining > 0: (3)
write_from_chrom = min(remaining, CHUNK_SIZE)
remaining -= write_from_chrom
chrom_calls = genomes[f"chromosome-{chrom}/calls"]
all_calls[all_start:all_start + write_from_chrom, :] = chrom_calls[
chrom_start: chrom_start + write_from_chrom, :]
all_start = all_start + write_from_chrom
(1) Первая позиция записи это значение номера блока, умноженная на размер блока.
(2) Мы проходим все размеры хромосом, пока не найдём с чего начать.
(3) Фрагмент может потребовать более одной хромосомы.
Не принимайте близко к сердцу если вы не понимаете полностью предыдущий код: этот код специфичен для своей области. Мы пытаемся работать по частям, что подходит для больших не умещающихся в оперативной памяти файлов.
Теперь мы можем воспользоваться простым пулом со множеством процессов с соответствием вызовов процессов каждому фрагменту.
from functools import partial
from multiprocessing import Pool
partial_process_chunk = partial(
process_chunk, genomes,
all_calls, chrom_sizes, CHUNK_SIZE)
def do_parallel():
with Pool() as p:
p.map(partial_process_chunk, range(all_calls.nchunks))
do_parallel()
Определяя partial_process_chunk, мы выполняем приложение частичной функции для упрощения вызовов
Pool.map. После этого мы применяем для обработки своего соответствия пул со множеством процессов. Более подробные сведения
об этом приводятся в Главе 3.
-
fsspec работает как универсальный интерфейс для хранилищ файлов, делая доступным для применения во множестве различных лежащих в их основе систем один и тот же API.
-
Поскольку при помощи fsspec у нас имеется универсальный API, заменять основу хранения значительно проще.
-
Хотя fsspec напрямую и не связан с производительностью, поверх него могут применяться различные библиотеки, включая Arrow и Zarr.
-
Parquet это формат хранения данных в столбцах, который делает возможным более эффективное хранение данных: данные обладают типами, потенциально могут сжиматься и организованы столбцами.
-
Parquet пользуется изощрёнными стратегиями кодирования данных, такими как словари или кодирование длиной последовательности, что делает возможным очень компактное представление, в особенности для данных с очевидными шаблонами и представлениями. Более того, этот формат расширяем, и в будущем могут появиться даже ещё более производительные расширения.
-
Parquet допускает разбиение данных на разделы, что снабжает программистов способностью одновременной обработки данных.
-
Наиболее распространённой методикой для работы с файлами размера больше оперативной памяти выступает фрагментация. Фрагментация поддерживается большим числом библиотек, включая pandas, Parquet и Zarr.
-
Zarr это современная библиотека обработки однородных многомерных массивов. Она возникла в мире Python и снабжена интерфейсами на основе NumPy.
-
Zarr поддерживает одновременную обработку сразу после установки. Сопровождение процессов параллельной записи является одним из тех, на которые следует обратить внимание, поскольку это не распространённая функциональная возможность для прочих библиотек.